
Eukarüootide ribosoom
EUKARÜOOTIDE RIBOSOOM
Programmeeritud valgusünteesi viib rakkudes läbi molekulaarne nanomasin – ribosoom. Ribosoomid on kõigis elusorganismides sarnase ehitusega ning nende ruumiline struktuur on evolutsiooniliselt kõrgelt konserveerunud. Ribosoomi põhistruktuuri moodustavad umbes 4400 RNA nukleotiidi (ribosomaalne RNA, rRNA) ning 33 ribosoomivalku (r-valk). Põhistruktuur sisaldab kõiki ribosoomi funktsionaalseid keskusi – dekodeeriv tsenter, peptidüültransferaasne tsenter, GTPaasidega seondumise piirkod, peptiidi väljumise tunneli algus ning A-, P- ja E-saidid kuhu seonduvad tRNA molekulid. Sellele vaatamata on valgusünteesi aparaat prokarüootides (eeltuumsed) ja eukarüootides (päristuumsed) erinev mitmes olulises aspektis. Eukarüootide ribosoomid on märkimisväärselt suuremad ning keerukama ehitusega. Lisandunud on rRNA järjestusi, r-valkude järjestusi ning uusi r-valke.

Pagaripärmi Saccharomyces cerevisiae ribosoomi 40S ja 60S alaühikute struktuur. Ribosomaalne RNA ja universaalselt konserveeritud ribosoomi valgud on tähistatud hallilt ja tumesiniselt. Arhede/eukarüootide spetsiifilised valgud on värvitud punaselt. Domeeni spetsiifilised valgud on tähistatud kollaselt. Näidatud on tRNA-de seondumise (A-, P- ja E-saidid) ligikaudne asukoht. Joonise tegemisel on kasutatud S. cerevisiae 80S ribosoomi 3D struktuuri (PDB koordinaadid 4V88) (Ben-Shem et al., 2011) ning programmi PyMol.
Arhede/eukarüootide spetsiifilised r-valgud ning nende poolt moodustunud alaühikuid ühendavad kontaktid.
Pagaripärmi ribosoom, nagu iga eukarüootne ribosoom, koosneb väikesest (40S) ja suurest (60S) ribosomaalsest alaühikust. Valgusünteesi initsiatsioonil moodustavad need alaühikud funktsionaalse 80S ribosoomi.
Alaühikute ühinemisel moodustuvad spetsiifilised kontaktid ehk alaühikute vahelised sillad. Pagaripärmi ribosoomi struktuurne mudel kirjeldab 17 silda. Nendest 12 silda on evolutsiooniliselt konserveerunud. Konserveerunud sillad asuvad ribosoomi põhistruktuuris ning sisaldavad RNA-RNA, RNA-valk ja valk-valk vahelisi kontaktide. Konserveerunud sildade funktsioone on põhjalikult uuritud. Need on olulised translatsiooni protsessiivsuse ja translatsiooni täpsuse tagamisel ning translokatsioonil ja ribosoomide rotatsiooni regulatsioonis.
Alaühikute vahelistest sildadest on viis silda spetsiifilised just eukarüootide ribosoomidele. Need sillad moodustuvad tänu RNA-valk ja valk-valk kontaktidele. Sildade tekkimisel on olulised ribosoomi eukarüoodi spetsiifilised struktuursed elemendid. Neli alaühikute vahelist silda (eB8, eB11, eB12 ja eB13) paiknevad ribosoomi perifeersetes regioonides. Ainult üks eukarüoodi-spetsiifiline sild, eB14, asub ribosoomi põhistruktuuris. Eukarüoodi spetsiifiliste sildade roll ribosoomi funktsionaalsuse jaoks on vähem uuritud. Meie poolt läbi viidud uuringud on esimesed süsteemsed analüüsid, et selgitada välja eukarüoodi spetsiifiliste alaühikute vaheliste sildade funktsioonid.
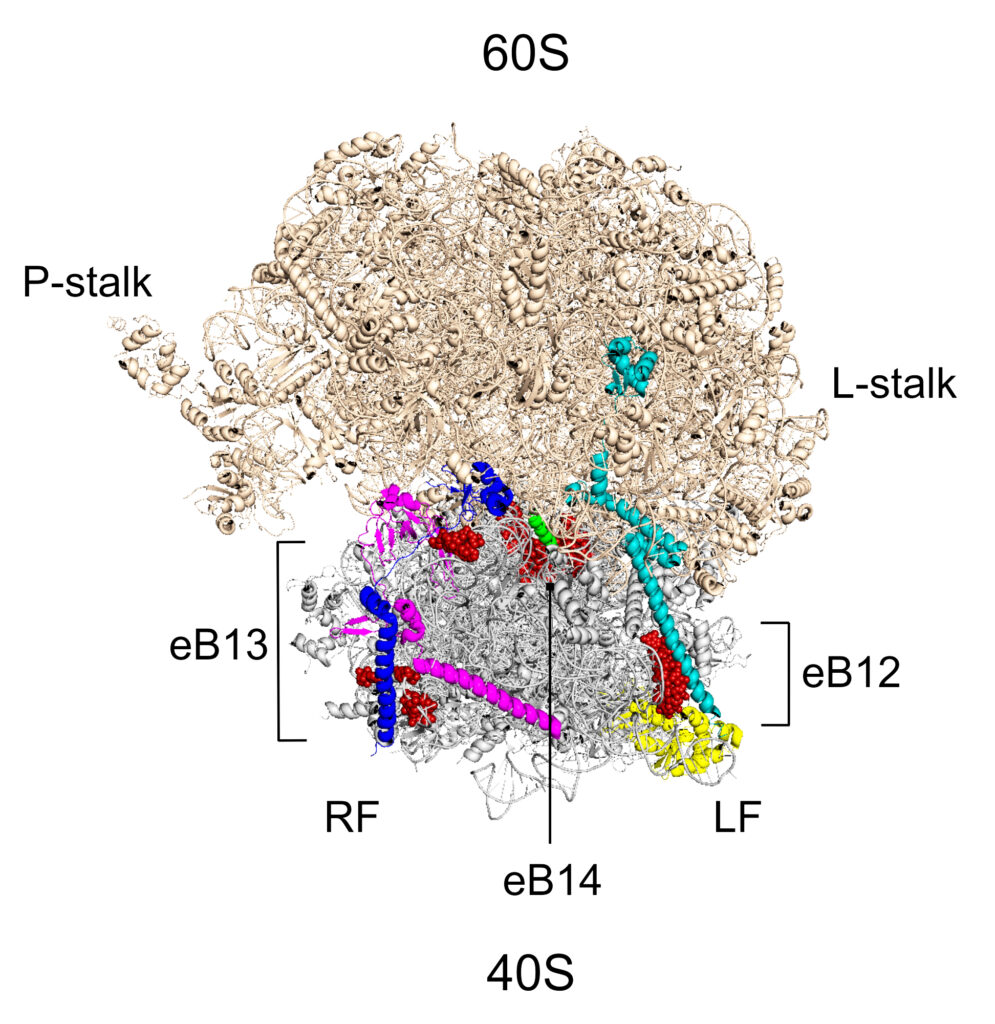
Pagaripärmi Saccharomyces cerevisiae 80S ribosoomi struktuur. Näidatud on suur alaühik (60S, helepruun) ja väike alaüksus (40S, helehall). Tähistatud on kolm eukarüootidele iseloomulikku alaühikute vahelist silda: eB12, eB13 ja eB14. Neid sildu moodustavad: ribosoomivalgud eL19 (helesinine), eL24 (tumesinine), eL41 (roheline), eS6 (lilla), eS7 (kollane) ja rRNA järjestused (punane). Joonise tegemisel on kasutatud S. cerevisiae 80S ribosoomi 3D struktuuri (PDB koordinaadid 4V88) (Ben-Shem et al., 2011) ning programmi PyMol.
Oma uurimistöös keskendume ribosoomi eukarüoodi spetsiifilisetele sildadele. Nende sildade olulisteks komponentideks on r-valgud, mis bakterites puuduvad. Seda spetsiifilist r-valkude rühma nimetatakse arhede/eukarüootide spetsiifilisteks (A/E-spetsiifilised) r-valkudeks (vt. põhjalikumat ülevaadet: Kisly ja Tamm, 2023). R-valgud eL19 ja eL24 koosnevad kolmest domeenist: N-terminaalne domeen, keskmine regioon ja C-terminaalne α-heelikaalne domeen. Mõlema valgu pikad C-terminaalsed domeenid osalevad alaühikute vaheliste eukarüoodi spetsiifiliste sildade eB12 ja eB13 moodustamisel.
Oleme konstrueerinud pärmitüved, mis ekspresseerivad r-valkude variante, milles puuduvad silla moodustamiseks vajalikud järjestused. Sillad eB12 ja eB13 on ülitähtsad pagaripärmi ribosoomi funktsionaalsuse jaoks (Kisly et al., 2016; Kisly et al., 2019). Need sillad tagavad ribosoomi alaühikute kokkusaamise, mis toimub translatsiooni initsiatsiooni etapis.
Valgu eL19 deletsioonimutantide analüüsil selgus, et eB12 silla funktsionaalsus sõltub valk-rRNA interaktsioonidest valgu eL19 ja 18S rRNA lisasegmendi ES6S vahel. Kuigi eB12 sild ei ole eluks hädavajalik, on see erakordselt oluline ribosoomi optimaalseks toimimiseks. Silla eB13 funktsioneerimise tagavad eelkõige valk-valk interaktsioonid valkude eL24 ja eS6 vahel.
Alaühikute vaheliste sildade kooperatiivsuse analüüsimiseks kasutasime pagaripärmi tüvesid, kus ribosoomides puuduvad erinevad sildade eB12, eB13 ja eB14 kombinatsioonid (Tamm et al, 2019). Näitasime, et need sillad ei funktsioneeri sõltumatult, vaid täidavad üksteist ning toetavad üheskoos ribosoomi funktsionaalsust.
Mutantsete ribosoomide iseloomustamiseks töötasime välja uue meetodi, mis põhineb rakuvabas translatsioonisüsteemis liitvalgu Renilla-Firefly sünteesil (Kisly et al., 2021). See meetod võimaldab määrata ribosoomide elongatsioonikiirust ja protsessiivsust ja seda sõltumatult valgusünteesi initsiatsiooni ja terminatsiooni etappidest. Lisaks saab määrata ribosoomide seiskumist spetsiifilistel mRNA järjestustel.
Käimasolevad projektid keskenduvad translatsiooni initsiatsiooni uurimisele. Analüüsime valgusünteesi alustamist spetsiaalsetelt RNA elementidelt (IRES, sisemine ribosoomi seondumispiirkond). Kilgi halvatuse viirus sisaldab IRES regiooni, mis töötab lisaks putukarakkudele ka pärmirakkudes. Seega on võimalik kasutada selle IRES-e uurimiseks Drosophila S2 rakuliini ning pagaripärmi erinevaid tüvesid. Analüüsime kuidas toimub IRES-e poolt algatatud valgusüntees mutantseid ribosoome sisaldavates rakkudes. Lisaks analüüsime translatsiooni initsiatsioonis olulise IRES RNA-80S ribosoomi kompleksi moodustumist kui puuduvad mõned ribosoomivalgud või ribosoomivalkude domeenid.
Modifitseeritud nukleotiidid ribosoomi RNAs
Ribosoomide RNA sisaldab lisaks tavapärastele nukleotiididel ka modifitseeritud nukleotiide. Need modifikatsioonid lisatakse rRNA molekulidesse pärast transkriptsiooni, ribosoomi küpsemise käigus. Kõige levinumad modifikatsioonid rRNA-s on riboosi 2’-O-metülatsioonid ja uridiini isomerisatsiooni tagajärjel tekkinud pseudouridiinid. Lisaks on kirjeldatud veel erinevaid lämmastikaluste modifikatsioone. Evolutsiooni käigus on modifitseeritud nukleotiidide hulk muutunud. Kui bakteri E. coli rRNA sisaldab 36 modifitseeritud positsiooni, siis pagaripärmi rRNA-s on neid juba üle 100 ning inimese rRNA-s üle 200. Evolutsiooni käigus on muutunud ka mehhanismid kuidas neid modifikatsioone rRNA-sse lisatakse. Bakteris viivad vastavaid reaktsioone läbi spetsiifilised ensüümid. Arhedes ja eukarüootides on lisaks spetsiifilistele ensüümidele kasutusel ka väikestel tuumakese-spetsiifilistel RNA-del (snoRNA) põhinev süsteem.
rRNA modifitseeritud nukleotiidid ei ole ühtlaselt ribosoomi struktuuris jaotunud vaid grupeeruvad ribosoomi funktsionaalselt olulistesse keskustesse: tRNA seondumise saidid, peptidüültransferaasi tsenter, dekodeerimise tsenter, peptiidi väljumise tunnel ja subühikute vahelised sillad. Bakteri E. coli, pagaripärmi S. cerevisiae ning inimese rRNAs paiknevate modifitseeritud nukleotiidide analüüsil selgus, et vaid üheksal juhul paiknevad need evolutsiooniliselt konserveerunud positsioonides. Neli positsiooni asuvad suure alaühiku peptidüültransferaasse tsentri piirkonnas.
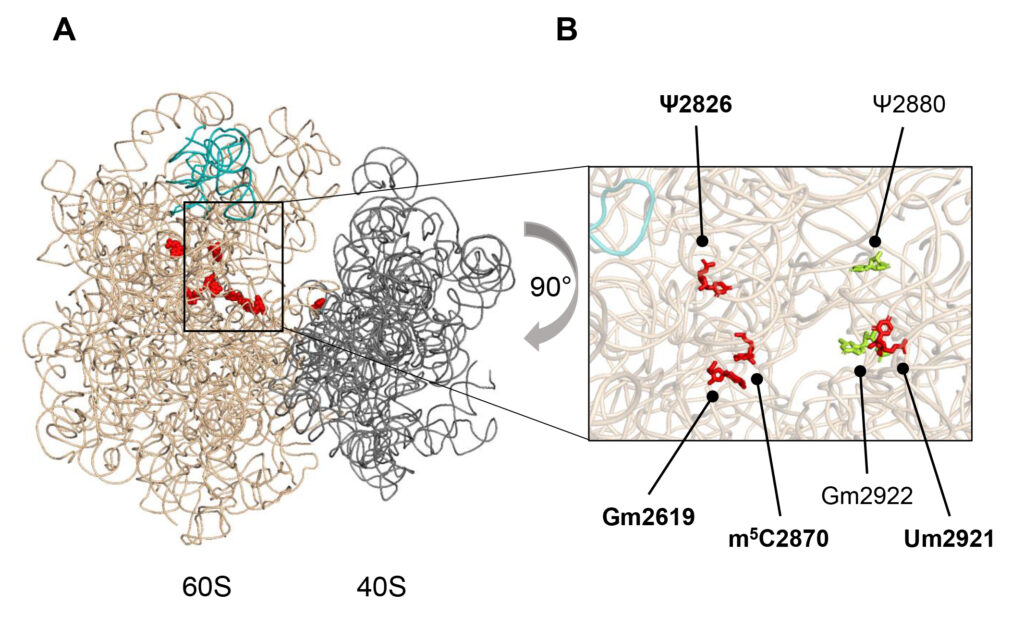
Pagaripärmi Saccharomyces cerevisiae 80S ribosoomi struktuur koos peptidüültransferaasse tsentri lähivaatega. Joonisel on näidatud rRNA modifitseeritud nukleotiidid, mille olulisust uuriti Leppik et al., 2024. Evolutsiooniliselt konserveerunud modifikatsioonid on värvitud punaseks.
Peptidüültransferaasse tsentri ümbruses paiknevate rRNA modifikatsioonide funktsiooni uurimiseks konstrueerisime pärmitüved, millel puudusid kolm snoRNA geeni ja/või mis ekspresseerisid kahe ribosoomi biogeneesis osaleva faktori ensümaatiliselt inaktiivseid variante (vt. teadusartiklit Leppik et al. 2024). Saadud pärmitüvi oli küll elujõuline kuid seitsme modifitseeritud nukleotiidi puudumine põhjustas rakkude kasvu aeglustumist ja külmatundlikkust. Lisaks toimus translatsioon mutantsetes rakkudes aeglasemalt ning täpsemalt. Seega tagab peptidüültransferaasses tsentris olev rRNA modifitseeritud nukleotiidide muster optimaalse ribosoomi aktiivsuse ning translatsiooni täpsuse.
Edasine uurimistöö keskendub evolutsiooniliselt konserveerunud modifitseeritud nukleotiidide olulisuse analüüsimisele. Kasutame konstrueeritud mutantseid pärmitüvesid ning viime nendesse tagasi funktsionaalsed snoRNA-d ning ensümaatiliselt aktiivsed biogeneesi faktorid. Analüüsime nendes tüvedes töötavate ribosoomide aktiivsust ning translatsioonilist täpsust. Selline ribosoomide „parandamise“ strateegia võimaldab selgitada üksikute modifitseeritud nukleotiide rolli ribosoomi funktsionaalsuse tagamisel.
KAITSTUD Doktoritööd
2019
Ivan Kisly
Juhendajad:
Tiina Tamm,
Jaanus Remme

