
Genoomikast ja selle arengusuundadest mittebioloogidele
7. Metagenoomika
Metagenoomika on DNA sekveneerimine ökosüsteemide tasemel. Alternatiivsed nimetused on veel keskkonnagenoomika ja ökogenoomika.

http://www.beach-backgrounds.com/bali-hai-stream-kauai-hawaii-beach-wallpaper/
Metagenoomide uurimiseks saadakse geneetiline materjal otse keskkonnaproovidest. Sealt sekveneeritakse spetsiaalne geen, milleks sageli on bakterite taksonoomilise jaotuse aluseks olev (s.t. liigiti erinev) 16S rRNA konserveerunud järjestus, et teha kindlaks elusorganismide (mikroobide) mitmekesisuse profiili.
Millises meditsiinis kasutatavas metoodikas sekveneeritakse samuti bakterite tuvastamiseks 16S rRNA järjestust?
Metagenoomika uuringud on näidanud, et tegelikult läheb mikroorganismide kultuuritingimustes kasvatamisega kaduma mikroobide looduslik mitmekesisus ja < 1% bakterite ning arhede liikidest jääb alles.
Lühidalt – metagenoomika baseerub kaasaegsetel genoomika tehnikatel (shotgun ja high – throughput sekveneerimine), et uurida mikroobide kooslust otse nende looduslikust keskkonnast, minnes mööda vajadusest isoleerida ja kultiveerida individuaalseid liike.
Metoodikad
Metagenoomika uuringutes kasutatakse laialt shotgun sekveneerimise metoodikat.
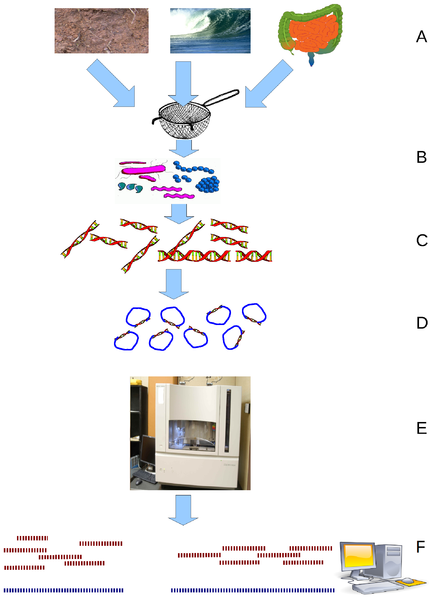
Skeem: Shotgun sekveneerimine metagenoomide uurimisel: A – proovide võtmine keskkonnast, B – osakeste filtreerimine (tavaliselt suuruse järgi), C – lüüsimine ja DNA ekstraheerimine, D – DNA kloonimine ja raamatukogu konstrueerimine, E – kloonide sekveneerimine, F – järjestuste assambleerimine kontigideks ja analüüs. http://en.wikipedia.org/wiki/File:Environmental_shotgun_sequencing.png
Metagenoomika kasutusvaldkonnad
Metagenoomikat saab kasutada reas valdkondades uute teadmiste hankimiseks:
- meditsiinis – Inimese Mikrobioomi projekti käigus viidi 250 uuritaval 15 – 18 kehapiirkonna mikroobikoosluse iseloomustamiseks läbi metabioomi sekveneerimine.
Teine projekt MetaHit (Metagenomics of Human Intestinal Tract) viidi läbi gastrointestinaalsete bakterite uurimiseks tervetel, ülekaalulistel ja ärritunud soole sündroomiga inimestel. Soole distaalse osa bakteritest ˃90% moodustasid 2 bakterite liiki Bacteroidetes ja Firmicutes. Geenisageduste alusel identifitseeriti 1244 metagenoomset klastrit, mis on olulised soolestiku tervise tagamisel. Neil bakteritel võis eristada kahte peamist funktsiooni:
-
-
-
-
-
- Housekeeping – mida vajatakse kõikjal organismis ja mis vastutavad peamiste metaboolsete radadae (nt. C metabolism) ja aminohapete tootmise eest.
- Soolestikuspetsiifiline funktsioon – adhesioon peremehe valkudega ja glükolipiidide suhkrujääkide eemaldamine.
-
-
-
-
Tulemusena leiti, et ärritunud soole sündroomiga patsientidel avaldus 25% vähem geene ja neil oli bakterite mitmekesisus väiksem.
Umbes pooled järjestustest oli võimalik reastada bakterite genoomsete järjestuste andmebaasi vastu, mis näitab, et paljud bakterite genoomid on veel uurimata.
Metagenoomika teised kasutusvõimalused:
- biokütuste tootmisel – mis sõltub mikroobidest, kes muudavad tselluloosi suhkruteks ja suhkrud etanooliks.
- biotehnoloogias ja põllumajanduses – 1g mulda sisaldab 10 9 – 10 10 mikroobi.
- ökoloogias – bakterid, kes transformeerivad ained bioloogiliselt aktiivsesse vormi.
Millistes valdkondades on veel võimalik kasutada metagenoomika tasemel lähenemist probleemide lahendamisel?
Kirjandus:


